NAR发表端粒序列多样性综合数据库TeloBase,覆盖超9000个物种
2023-09-08 测序中国 网络 发表于上海
研究团队创建了TeloBase数据库。该数据库包含9000多个物种的端粒序列,不仅提供交互式操作和数据可视化的图形,还允许基于用户的管理,避免了今后数据更新对管理员的过度依赖,以确保数据库更新的及时性
导读
端粒是一种复杂的核蛋白结构,可以区分染色体的自然末端和DNA断裂,并保护基因组的编码区不因染色体末端的不完全复制而丢失。在哺乳动物和植物中,端粒酶的活性在发育过程中受到严格调控,并且仅限于具有高增殖能力的组织。端粒DNA通常由短序列重复的基序组成,从广义上说,这些基序遵循一定的规律。但在真实世界中,端粒的真正差异比最初想象的要多得多。
自发现第一个端粒序列(TTGGGG)以来,已经过去了40多年。期间人们发现了酵母端粒序列的多样性,也建立了主要分类“规范”端粒序列的基本观点。目前,端粒酶数据库和植物rDNA数据库均提供了有关端粒序列多样性的信息。植物rDNA数据库主要关注rDNA及其染色体的位置和排列;相比之下,端粒酶数据库包含植物界以外其他类群的信息。但目前还没有一个数据库可以覆盖关于端粒的所有信息。
为了填补这一空白,来自捷克马萨里克大学等单位的研究团队经过文献检索,并从NCBI数据库中公开可得的NGS数据中进一步搜索潜在的端粒序列,扩大了目前已知端粒序列的物种数量。为了高效利用所有的可用数据,研究团队创建了TeloBase数据库。该数据库包含9000多个物种的端粒序列,不仅提供交互式操作和数据可视化的图形,还允许基于用户的管理,避免了今后数据更新对管理员的过度依赖,以确保数据库更新的及时性。该研究发表在Nucleic Acids Research上,文章题为“TeloBase: a community-curated database of telomere sequences across the tree of life”。

文章发表在Nucleic Acids Research
首先,研究人员使用谷歌学术收集了大量与端粒序列相关的数据,并对相关文献进行了系统的综述。虽然有大量的搜索条目,但其中很多并非同行评议文章。最终,研究团队通过文献检索发现了1619篇相关论文,这比在植物rDNA数据库和端粒酶数据库中引用的文献分别高20和26倍。
为了增加目前已知或预测的端粒序列的物种数量,研究人员在SRA库(NCBI)中存储的原始测序数据中寻找候选端粒数据,利用串联重复查找分析(TRFi),在数据集内增加了136%的物种。相比之下,文献搜索识别出2940个处于实验状态的物种和889个处于模型状态的物种。来自原始数据搜索的另外810个物种与来自文献搜索的结果相匹配。这些额外的数据可以进一步证实和验证已知的序列。通过这两种方法鉴定出的大部分端粒序列属于动物界和植物界。
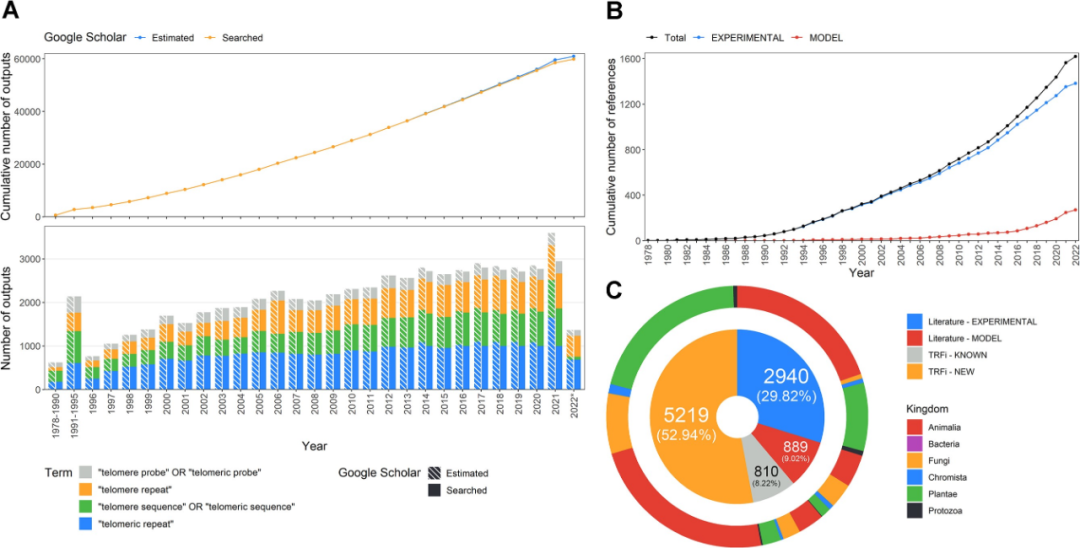
图1. 数据收集的汇总统计
为了更好地使用该数据集,研究团队开发了数据库TeloBase,包含了9000多个物种的端粒序列。
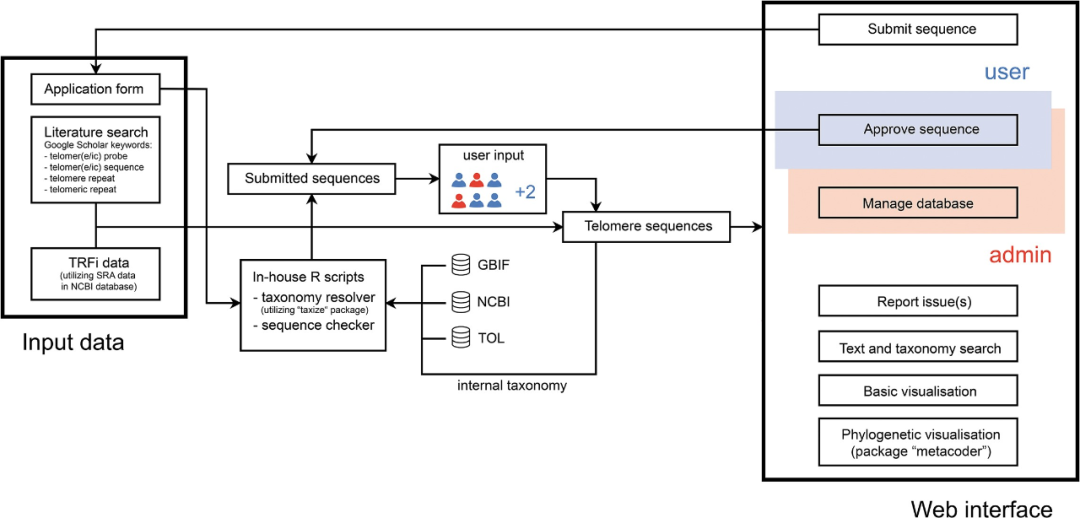
图2. TeloBase数据库原理图
1.输入数据,即由研究人员新提交或管理员新收集的数据,存储在“端粒数据标签”。每个条目都提供了名称、序列、位置、状态、出版物或NCBI的链接。
2.基于可视化的端粒序列分布在一个各自的分类单元。TeloBase使用户能够以热树图的形式为选定的端粒序列分布着色。由于计算的限制,只绘制了从所选分类名称开始的两个递减分类等级,这一功能有助于分析端粒序列中普遍存在的潜在关联。
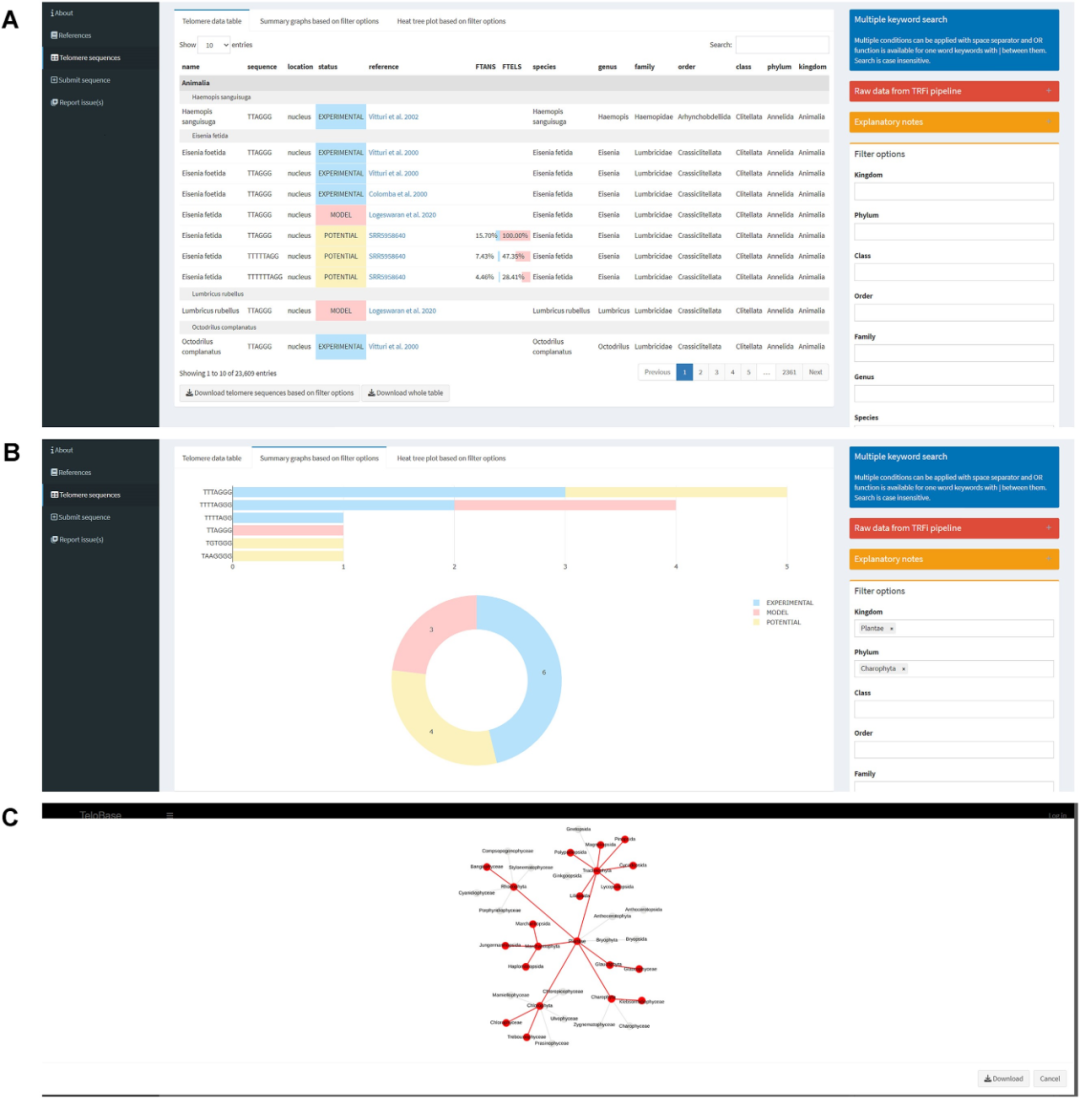
图3. TeloBase操作界面图。
3.端粒新序列的应用及序列管理。添加到TeloBase的其他条目不依赖于管理员,任何用户都可以在注册后对提交的条目进行查看后添加。为了加快提交过程,可以根据内部分类信息和外部分类信息的组合自动填写分类信息。TeloBase还自动将输入的序列转换为数据库中已经存在的迭代序列。
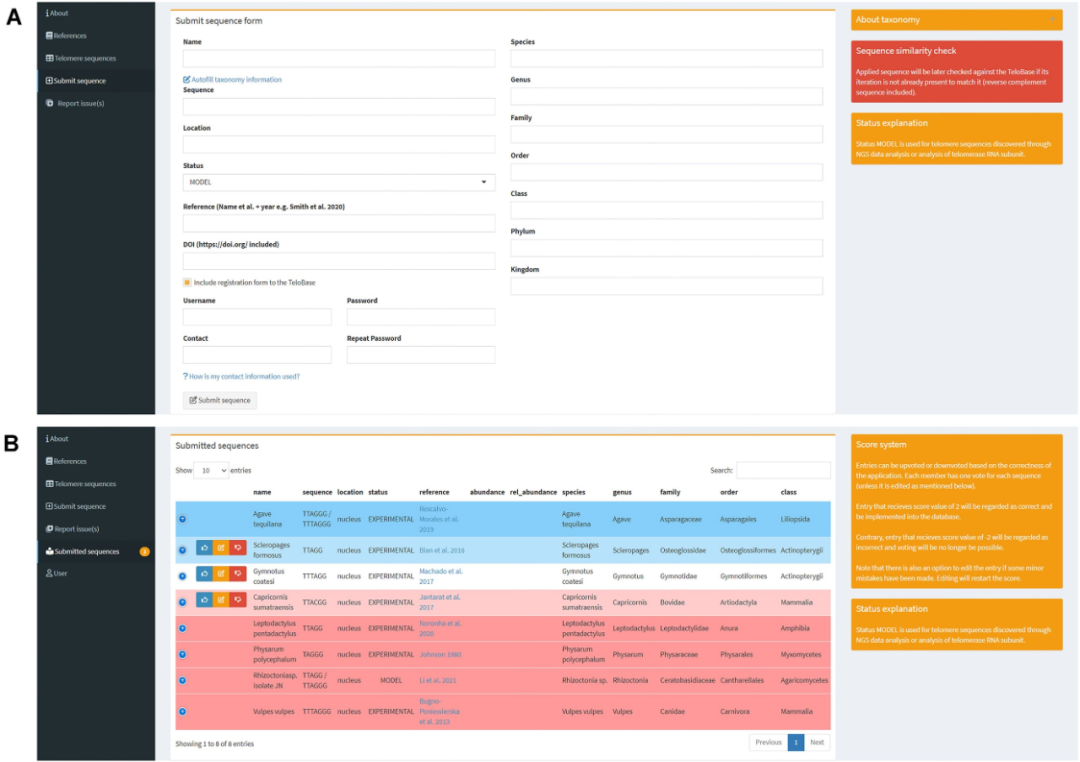
图4. 端粒新序列的应用及管理
研究团队利用曲霉属证明了TeloBase的有效性,其中NGS数据分析中具有潜在状态的端粒序列在物种之间显示出潜在的巨大多样性。此外,研究团队在金壳果科科的维管植物中发现了一种新的端粒序列(TTTATTAGGG)。以上结果显示TeloBase为端粒多样性带来了新的见解。
综上所述,该团队从文献和NGS数据搜索中构建了一个相对完整的端粒序列数据集,并在TeloBase数据库中实现应用。TeloBase允许数据的交互操作和可视化,以及对新条目的简单应用和管理。基于这些特征,研究人员认为TeloBase能够为未来端粒序列多样性和端粒进化提供可靠和可行的信息来源,有望带来深入了解,特别是由于可用测序数据的数量不断增加。
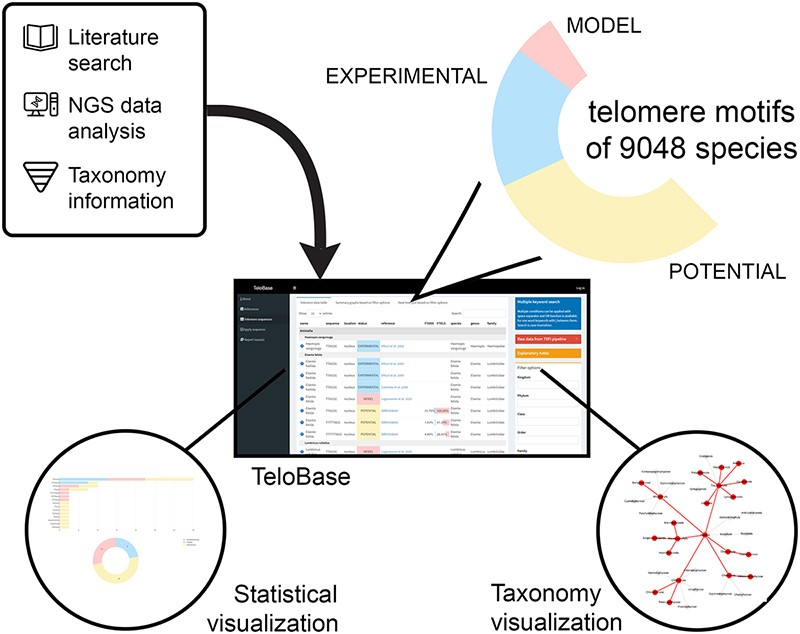
图5.TeloBase数据库的内容和功能总结
TeloBase数据库:
http://cfb.ceitec.muni.cz/telobase/
参考资料:
Martin Lyčka., et al. TeloBase: a community-curated database of telomere sequences across the tree of life, Nucleic Acids Research, 2023;, gkad672.
https://academic.oup.com/nar/advance-article/doi/10.1093/nar/gkad672/7246534
本网站所有内容来源注明为“williamhill asia 医学”或“MedSci原创”的文字、图片和音视频资料,版权均属于williamhill asia 医学所有。非经授权,任何媒体、网站或个人不得转载,授权转载时须注明来源为“williamhill asia 医学”。其它来源的文章系转载文章,或“williamhill asia 号”自媒体发布的文章,仅系出于传递更多信息之目的,本站仅负责审核内容合规,其内容不代表本站立场,本站不负责内容的准确性和版权。如果存在侵权、或不希望被转载的媒体或个人可与williamhill asia 联系,williamhill asia 将立即进行删除处理。
在此留言







