Nature Microbiology:使用嵌套PCR和纳米孔测序检测敏感脊髓灰质炎病毒
2023-08-21 Jenny Ou MedSci原创 发表于上海
需要进一步评估,以比较检测的准确性和这两种方法生成VP1序列的速度,同时考虑实施的难易性和员工培训要求。
尽管全球消除脊髓灰质炎倡议(GPEI)自1988年成立以来取得了实质性进展,但在疫苗接种覆盖率低的国家,脊髓灰质炎仍然是一个主要的公共卫生问题。在脊髓灰质炎病毒爆发期间,使用口服脊髓灰质炎病毒疫苗(OPV)进行大规模疫苗接种活动,以阻止传播。然而,大便样本运输缓慢、使用细胞培养进行耗时的病毒隔离和测序能力不足相结合,推迟了疫情反应,并减少了大规模疫苗接种运动的影响。

2020年8月,非洲地区被宣布中断了野生脊髓灰质炎病毒(WPV)的传播。OPV疫苗接种导致了循环疫苗衍生脊髓灰质炎病毒(cVDPV)的爆发,这是通过恢复活疫苗菌株的减弱突变而发生的。接种疫苗后,活疫苗菌株在粪便中脱落,并可能传播到免疫不足的人群中,衰减的突变会随着时间的推移而消失。
在WPV后时代,幼儿的血清型2(cVDPV2)流行病困扰着非洲和西亚。2020年,27个国家报告了959例由cVDPVV2引起的瘫痪病例,包括非洲21个国家;2021年,全球报告了692例由cVDPV2引起的病例和20例由1血清型cVDPV引起的病例,主要在非洲,包括尼日利亚和刚果民主共和国(DRC)。2022年,至少报告了843例VDPV病例,其中502例在DRC。
在刚果民主共和国,在最后一例WPV病例发生10年后,由于在使用血清型2 OPV以应对现有疫情后出现cVDPV2,cVDPV2几乎持续传播。监测不足和疫情确认前的时间过长,阻碍了应对。脊髓灰质炎病毒监测基于从急性弛缓性麻痹(AFP)儿童及其接触者那里收集大便样本,以及环境(污水)采样。有效的脊髓灰质炎病毒监测依赖于高质量的样本收集和实验室测试。从AFP病例中收集的粪便被认为足以用于测试,如果两个粪便间隔48小时,在瘫痪开始后2周内收集,并通过冷链到达并附有适当的文件。
在刚果民主共和国,2018年,AFP大便样本收集不足的病例比例为23%。此外,样品运往实验室、样品实验室测试和国际运往(运往南非)测序的后勤挑战导致脊髓灰质炎病毒爆发检测延迟。由于这些后勤问题,估计每增加一周(非洲地区的平均数据)爆发的病例数将增加约12%(95%可信间隔5-21%)。世界卫生组织(WHO)认为,延迟检测是根除脊髓灰质炎计划面临的主要挑战之一。
通过实施直接分子检测和纳米孔测序(DDNS)方法,可以实现检测和响应方面的这些改进。DDNS将粪便样本的快速、直接检测与现场测序相结合,避免了样品的国际运输,并能够快速应对疫情。它可以在任何已经使用PCR的实验室实施,包括INRB,其中Illumina和纳米孔测序已用于埃博拉病毒、麻疹、猴痘和严重急性呼吸综合征冠状病毒。
2023年8月17日发表在Nature Microbiology的研究,为了验证DDNS的实施,以便将其视为世卫组织全球脊髓灰质炎实验室网络(GPLN)的推荐方法,研究人员在刚果民主共和国进行了一项前瞻性研究,以评估DDNS协议的应用,并将其与脊髓灰质炎病毒监测的黄金标准细胞培养方法进行比较。
研究人员报告了DDNS与细胞培养的敏感性和特异性、测序准确性、实验室花费的时间和相关成本数据。本研究报告了2021年8月10日至2022年2月4日期间刚果民主共和国疑似脊髓灰质炎病例及其接触者的粪便样本的预期测试。
DDNS在62/2,339(2.7%)的样本中检测到脊髓灰质炎病毒,而细胞培养、定量PCR和桑格测序的金标准组合在51/2,339(2.2%)的相同样本中检测到脊髓灰质炎病毒。在常规监控条件下,DDNS在7天内(中位数)提供了病例确认。DDNS使三种2型循环疫苗衍生脊髓灰质炎病毒爆发的确认比金本位制方法早23天(平均)早23天(范围为6-30天)。通过这两种方法获得的序列之间的平均序列相似性为99.98%。本文数据证实了在国家脊髓灰质炎病毒实验室实施DDNS的可行性。
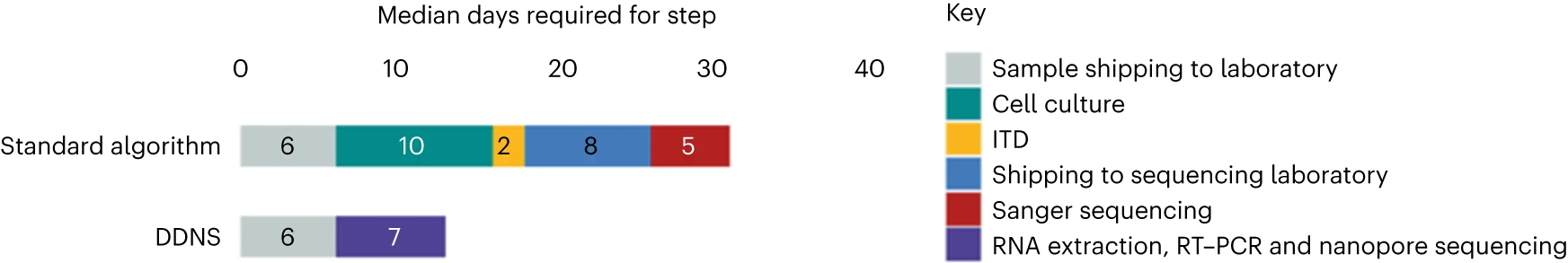
27个cVDPV2阳性大便样本的两个协议中每个诊断步骤所需的中位时间
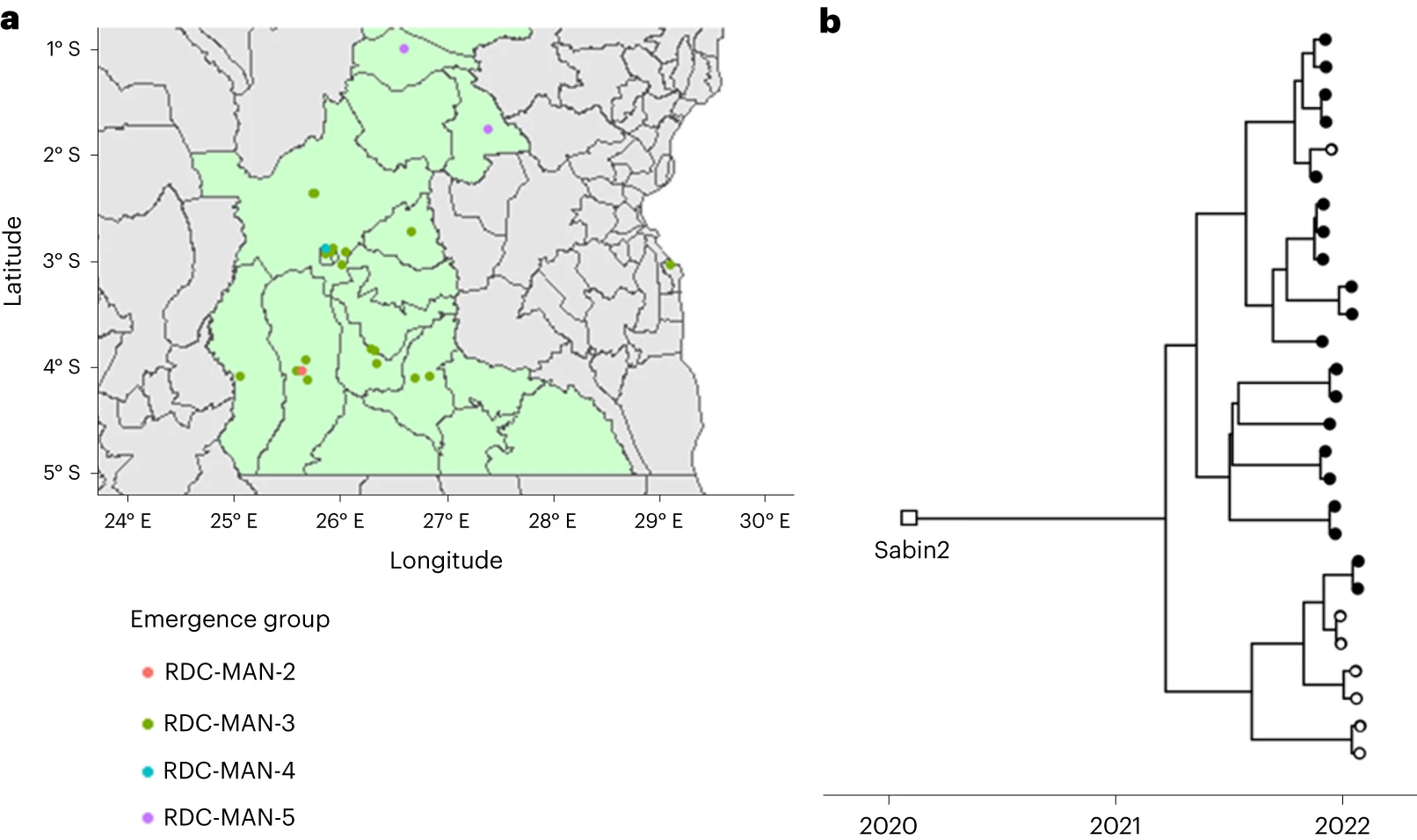
DDNS检测到的cVDPV2爆发
本文报告了一项前瞻性验证研究,该研究将DDNS与在刚果民主共和国大便样本中检测脊髓灰质炎病毒的金标准检测方法进行了比较。本文数据证实,DDNS可以作为刚果民主共和国快速、敏感和具有成本效益的监控工具;在国家脊髓灰质炎病毒实验室实施DDNS是可行的。虽然在统计上没有动力与金本位检测方法进行直接比较,但本文表明,DDNS在检测脊髓灰质炎病毒方面至少与培养物一样敏感。
原始出处
Shaw, A.G., Mampuela, T.K., Lofiko, E.L. et al. Sensitive poliovirus detection using nested PCR and nanopore sequencing: a prospective validation study. Nat Microbiol (2023). https://doi.org/10.1038/s41564-023-01453-4
本网站所有内容来源注明为“williamhill asia 医学”或“MedSci原创”的文字、图片和音视频资料,版权均属于williamhill asia 医学所有。非经授权,任何媒体、网站或个人不得转载,授权转载时须注明来源为“williamhill asia 医学”。其它来源的文章系转载文章,或“williamhill asia 号”自媒体发布的文章,仅系出于传递更多信息之目的,本站仅负责审核内容合规,其内容不代表本站立场,本站不负责内容的准确性和版权。如果存在侵权、或不希望被转载的媒体或个人可与williamhill asia 联系,williamhill asia 将立即进行删除处理。
在此留言









