【期刊导读】预测聚乙二醇干扰素α治疗应答的潜在生物标志物:CXCR4_rs28367495
2024-04-18 雨露肝霖 雨露肝霖 发表于陕西省
TRIM26_rs116806878及BST2_rs9576均是预测PegIFNα治疗应答的潜在生物标志物。
编者按
近年来,关于单核苷酸多态性(SNP)与聚乙二醇干扰素α(PegIFNα)的治疗应答密切相关的证据越来越丰富,已有研究证实TRIM22 SNP rs10838543 CC基因型,以及SNP rs7519753-C等位基因能较好预测PegIFNα的治疗应答;南方医科大学南方医院的蒋德科教授团队也证实,TRIM26_rs116806878及BST2_rs9576均是预测PegIFNα治疗应答的潜在生物标志物。
近期,侯金林教授同蒋德科教授团队再次发表相关研究,共筛选出22个趋化因子受体4 (CXCR4)相关的SNPs,其中携带CXCR4_rs28367495 CC等位基因的HBeAg阳性慢乙肝患者经PegIFNα治疗后,其联合应答(CR)率更高,HBsAg水平更低,HBsAg降幅更大。研究同时还发现结合CXCR4_rs28367495与另外5个SNPs(既往已报道的可预测PegIFNα治疗应答的预测因子)的多基因评分(PGS)能更好地预测PegIFNα的治疗应答。

研究方法
一项回顾性队列研究,纳入945例HBeAg阳性慢乙肝患者,所有患者均接受PegIFNα治疗至少48周,随访24周。分为两个独立队列,队列1纳入2010年4月至10月全国11家医院的238例慢乙肝患者,队列2纳入2013年1月至2015年8月全国43家医院或临床中心的707例慢乙肝患者。纳入标准:年龄18 - 65岁、HBsAg阳性至少6个月、HBeAg阳性、HBV DNA > 105 copies/mL、2 ≤ ALT < 10 × 正常值上限(ULN)、入组时无肝硬化、6个月内未接受过抗病毒治疗。
主要疗效终点为HBeAg血清学转换、平均HBV DNA水平、HBV DNA降幅、联合应答(CR)率、HBsAg水平及HBsAg降幅。联合应答(CR)定义为HBeAg血清学转换且HBV DNA < 3.3 log10 IU/mL。
患者特征
在队列1和队列2中,超70%为男性患者,平均年龄约29岁,HBV基因型为B/C型的患者比例分别为99.2%和96.2%,汉族分别占98.7%和92.9%,为排除遗传因素的影响,后续仅分析汉族患者。两组患者的HBV DNA水平相似(P = 0.135);但队列1的HBsAg水平和HBeAg水平均显著低于队列2(P 均< 0.001),ALT水平显著高于队列2(P = 0.029)。在两个队列中,与PegIFNα治疗应答相关的5个SNPs(STAT4_rs7574865、CFB_rs12614、GNLY_rs11127、CD55_ rs28371597和TRIM26_rs116806878)的分布相似(P 均> 0.05)。
接受PegIFNα治疗的中国HBeAg阳性乙肝患者的临床特征

研究结果
一.CXCR4_rs28367495 CC等位基因能更好预测HBeAg阳性慢乙肝患者对PegIFNα的治疗应答
本研究共筛选出22个CXCR4相关的标签SNPs(tag SNPs),进一步分析发现CXCR4_rs28367495的C等位基因与PegIFNα的治疗应答显著相关。随着rs28367495 C等位基因的增加(TT, 0; CT, 1; CC, 2),慢乙肝患者的HBeAg血清学转换率不断升高,平均HBV DNA水平逐渐下降,HBV DNA降幅逐渐升高,随着治疗时间和随访时间的延长,相关差异更为显著。
1 携带CXCR4_rs28367495 CC等位基因的患者经PegIFNα治疗后的HBeAg血清学转换率及HBV DNA降幅显著更高
例如在队列1+2中,与TT基因型和CT基因型患者相比,CC基因型患者经PegIFNα治疗后,第72周时的HBeAg血清学转换率(P = 0.001,图1 C)及HBV DNA降幅显著升高(P = 0.007,图1 I),平均HBV DNA水平显著降低(P = 0.002,图1 F)。
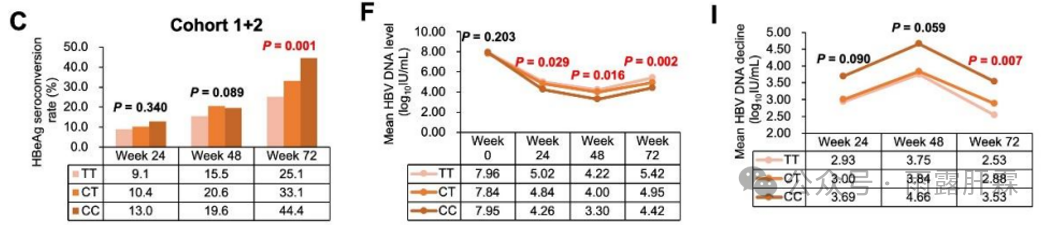
图1 在队列1+2中,HBeAg阳性慢乙肝患者经PegIFNα治疗后各时间点时,CXCR4_rs28367495的各基因型与HBeAg血清学转换率(C)、HBV DNA水平(F)和HBV DNA降幅(I)的关系比较
2 携带CXCR4_rs28367495 CC等位基因的患者PegIFNα治疗后的综合应答率显著更高
CXCR4_rs28367495 的C等位基因越多的患者(CC基因型 vs. CT基因型 vs. TT基因型)在72周时的CR率显著更高:队列1(36.4% vs. 33.3% vs. 17.5%; P = 0.008)、队列2(32.4% vs. 23.2% vs. 16.1%; P = 0.004)和队列1+2(33.3% vs. 25.7% vs. 16.5%; P = 1.51 × 10-4)(图2 A-C)。多因素logistic回归分析证实,CXCR4_rs28367495与各队列72周时的CR率显著相关(各队列的P 均< 0.05)。

图2 队列1(A)、队列2(B)和队列1+2(C)慢乙肝患者经PegIFNα治疗后各时间点,CXCR4_rs28367495的各基因型(TT、CT、CC)与联合应答率的关系比较
3 携带CXCR4_rs28367495 CC等位基因的患者经PegIFNα治疗后的HBsAg降幅更高
评估CXCR4_rs28367495与HBsAg水平和HBsAg降幅的关系时发现,在队列2和队列1+2中,与TT或CT基因型患者相比,CC基因型患者在PegIFNα治疗后的第24周、48周和72周时的平均HBsAg水平显著更低(P 均< 0.05),HBsAg降幅显著更高(P 均< 0.05)(图2 D-I)。多因素logistic回归分析表明,在队列1+2中CXCR4_rs28367495基因型与患者经PegIFNα治疗后72周时的平均HBsAg水平(P = 0.003)和HBsAg降幅(P = 0.003)呈显著正相关。
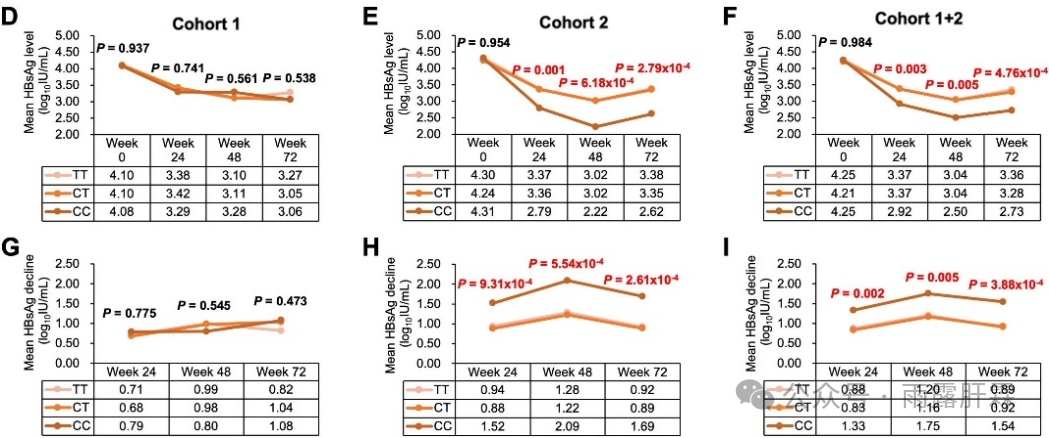
图2 D-I 各队列慢乙肝患者经PegIFNα治疗后各时间点时,CXCR4_rs28367495的各基因型(TT、CT、CC)与HBsAg水平(D-F)及HBsAg降幅(G-I)的关系比较
二.构建的多基因评分(PGS)能更准确地预测PegIFNα的治疗应答
1 PGS值越大,患者经PegIFNα治疗后的HBeAg血清学转换率及HBV DNA降幅越高
既往研究证实,STAT4_rs7574865、CFB_rs12614、GNLY_rs11127、CD55_rs28371597、TRIM26_rs116806878这5个SNPs均可预测PegIFNα的治疗应答,本研究在此基础上还纳入了CXCR4_rs28367495,构建了新的PGS模型,以期更好地预测HBeAg阳性慢乙肝患者对PegIFNα的治疗应答。
结果发现患者的PGS值越大,患者对PegIFNα的治疗应答率越高,尤其是在较长时间的治疗和随访中。例如,各队列中PGS值较高的患者在72周时的HBeAg血清转换率显著更高(P 均< 0.05; 图3 A-C),HBV DNA水平显著更低(P 均< 0.05; 图3 D-F),HBV DNA降幅显著更大(P 均< 0.05; 图3 G-I)。
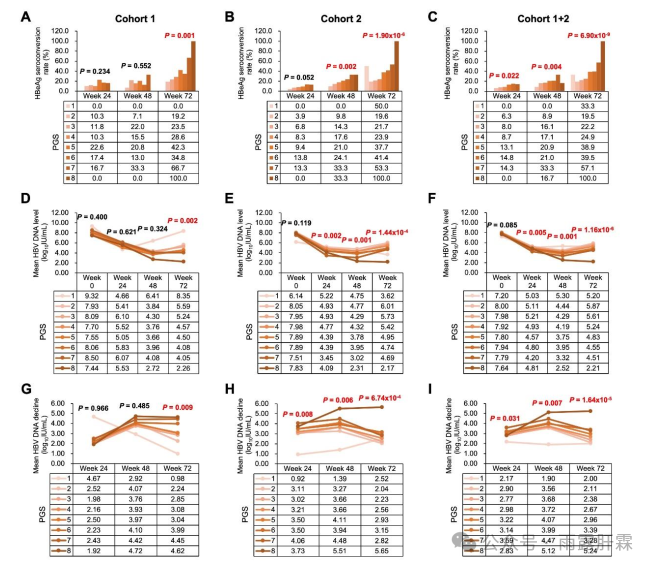
图3 各队列中,PGS值与HBeAg阳性慢乙肝患者经PegIFNα治疗后的HBeAg血清学转换率(A-C)、HBV DNA水平(D-F)及HBV DNA降幅(G-I)的关系
2 PGS值越大,患者经PegIFNα治疗后的CR率越高
在72周时,随着PGS值的升高(1→8),各队列患者的CR率不断提高(图4 A-C):
-
队列1(0.0%、3.8%、13.7%、23.2%、36.5%、30.4%、66.7%和100.0%;P = 3.45 × 10-6);
-
队列2(0.0%、9.8%、11.2%、17.6%、29.7%、31.0%、40.0%和100.0%;P = 8.21 × 10-9);
-
队列1+2 (0.0%、7.8%、11.8%、18.8%、31.6%、30.9%、47.6%和100.0%;P = 1.26 × 10-13)。
多因素logistic回归分析表明,在各队列中,PGS值均与PegIFNα治疗后72周时的CR率显著相关(各队列P 均< 0.05)。
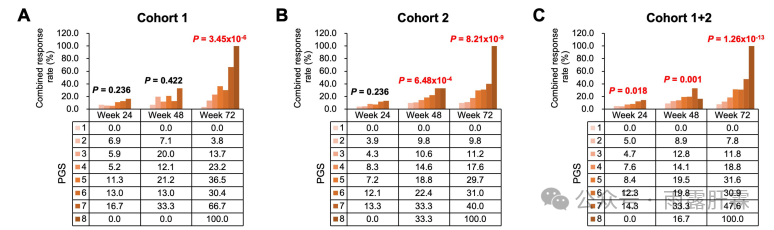
图4 A-C 各队列中,PGS与HBeAg阳性慢乙肝患者经PegIFNα治疗后CR的关系
3 PGS值越大,患者经PegIFNα治疗后的HBsAg降幅越大
患者的PGS值越高,平均HBsAg水平越低(图4 D-F),HBsAg降幅越大(图4 G-I)。例如,在队列1+2中,PGS值为1→8时,患者在第72周时的HBsAg水平显著降低 (P = 4.90 × 10-4;图3 F),HBsAg降幅显著增加(P = 0.005;图4 I)。多因素logistic回归分析表明,在队列1+2中,PGS值与HBsAg水平和HBsAg降幅均呈显著正相关(P = 0.004)。
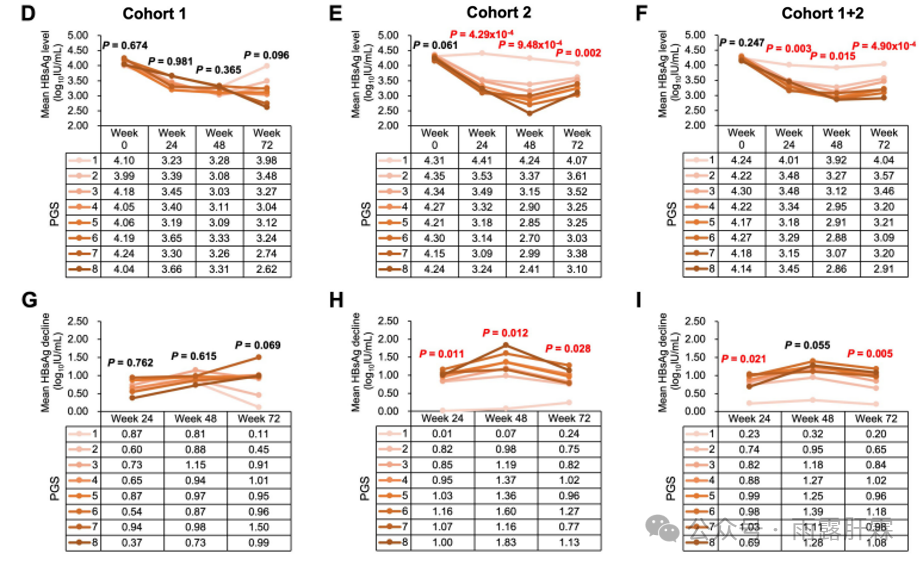
图4 各队列中,PGS值与HBeAg阳性慢乙肝患者经PegIFNα治疗后的HBsAg水平(D-F)及HBsAg降幅(G-I)的关系
三.ROC分析:CXCR4_rs28367495及PGS模型均能较好预测PegIFNα的治疗应答
通过ROC分析评估所有变量预测PegIFNα治疗应答的能力,结果显示在单个预测因子中,与年龄、HBV基因型和ALT相比,CXCR4_rs28367495及PGS的受试者工作特征曲线下面积(AUC)均更大。此外,整合模型的预测性能最佳,其AUC为0.724(图5)。
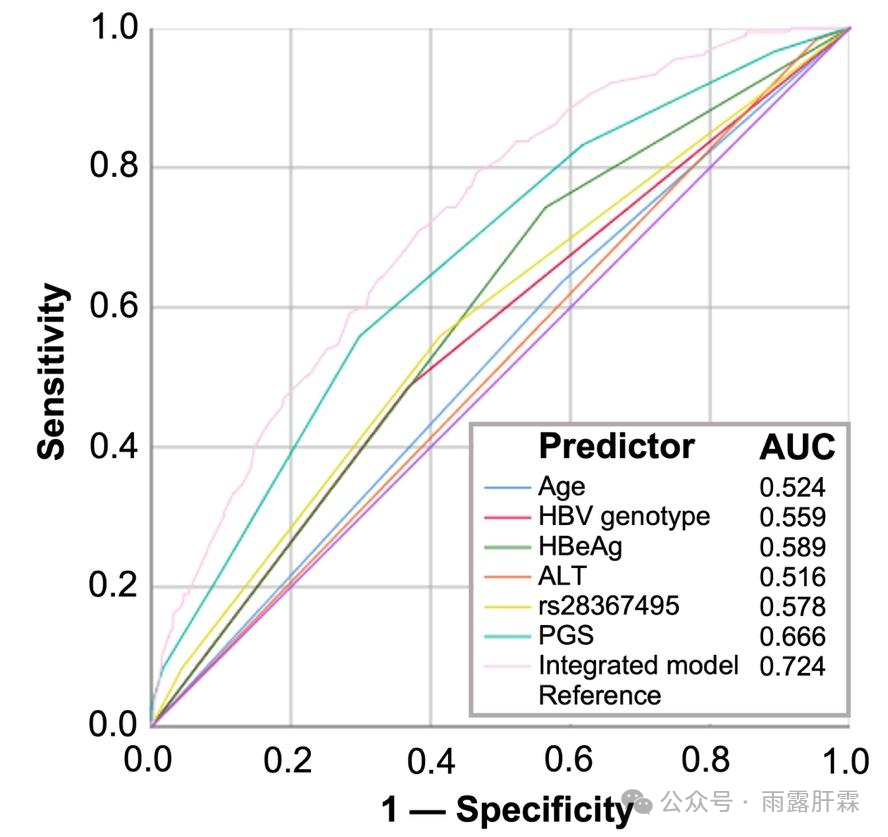
图5 预测变量与整合模型预测HBeAg阳性慢乙肝患者对PegIFNα治疗应答的AUROC
肝霖君有话说
CXCR4是一种α趋化因子受体,可通过不同的途径激活多种生物学过程,包括多种细胞功能及生理和免疫过程。本研究回顾性分析发现携带CXCR4_rs28367495 CC基因型的慢乙肝患者对PegIFNα治疗应答更好,该SNP或可成为预测PegIFNα治疗应答的预测指标。
目前已有多个PGS模型用于预测PegIFNα治疗HBeAg阳性慢乙肝患者的疗效,本研究通过整合6个SNPs(包括本研究新发现的CXCR4_rs28367495以及5个既往证实可预测PegIFNα治疗应答的SNPs)构建了一个新的PGS模型,该模型能更好地预测PegIFNα的治疗应答,这也为PegIFNα的治疗策略优化提供了新的方向,当然相关研究在未来仍需要进一步的探索。
参考文献:
Luo M, Dong C, Liang X, et al. A genetic variant of CXCR4 predicts pegylated interferon-alpha treatment response in HBeAg-positive chronic hepatitis B patients[J]. J Clin Microbiol. 2024, 62(2): e0139623.

本网站所有内容来源注明为“williamhill asia 医学”或“MedSci原创”的文字、图片和音视频资料,版权均属于williamhill asia 医学所有。非经授权,任何媒体、网站或个人不得转载,授权转载时须注明来源为“williamhill asia 医学”。其它来源的文章系转载文章,或“williamhill asia 号”自媒体发布的文章,仅系出于传递更多信息之目的,本站仅负责审核内容合规,其内容不代表本站立场,本站不负责内容的准确性和版权。如果存在侵权、或不希望被转载的媒体或个人可与williamhill asia 联系,williamhill asia 将立即进行删除处理。
在此留言
















#生物标志物# #聚乙二醇干扰素α# #CXCR4_rs28367495#
38